La diabetes tipo 1 (DT1) es una enfermedad multifactorial que resulta de una compleja interacción de factores genéticos y ambientales, llevando a la destrucción autoinmune de las células beta en el páncreas. Aunque no sigue un patrón de herencia mendeliana, presenta un claro componente genético que predispone a ciertos individuos a la enfermedad. La DT1 involucra múltiples tipos celulares, como linfocitos T autorreactivos que atacan las células beta, y diversas células inmunitarias y no inmunitarias del páncreas. Esta complejidad genética y celular exige un enfoque integral para entender los mecanismos patogénicos y desarrollar estrategias efectivas de diagnóstico precoz y tratamiento.
Las ciencias ómicas estudian conjuntos completos de moléculas dentro de un organismo, como la transcriptómica (ARN) y la proteómica (proteínas), ofreciendo una visión holística de las redes biológicas y cómo estas moléculas interactúan y regulan procesos biológicos. La genómica, por su parte, se centra en el estudio del genoma completo de un organismo, incluyendo la secuenciación, análisis e interpretación de su material genético. Este campo abarca la identificación de genes y sus secuencias, así como su contribución a la estructura, función y evolución del genoma.
Entender mejor los aspectos genómicos relacionados con la DT1 ofrece varios beneficios clave. Permite un diagnóstico temprano al identificar marcadores genéticos asociados con la predisposición a la enfermedad, facilitando la monitorización y la intervención precoz. Además, los avances en genómica posibilitan la medicina personalizada, ajustando el tratamiento según el perfil genético de cada paciente para así mejorar la eficacia y reducir los efectos secundarios. Finalmente, una comprensión profunda de la base genética de la DT1 impulsa el desarrollo de nuevas terapias al identificar dianas específicas para la intervención y diseñar tratamientos que aborden las alteraciones genéticas subyacentes.
Técnicas para el estudio del genoma de células únicas (single cell)
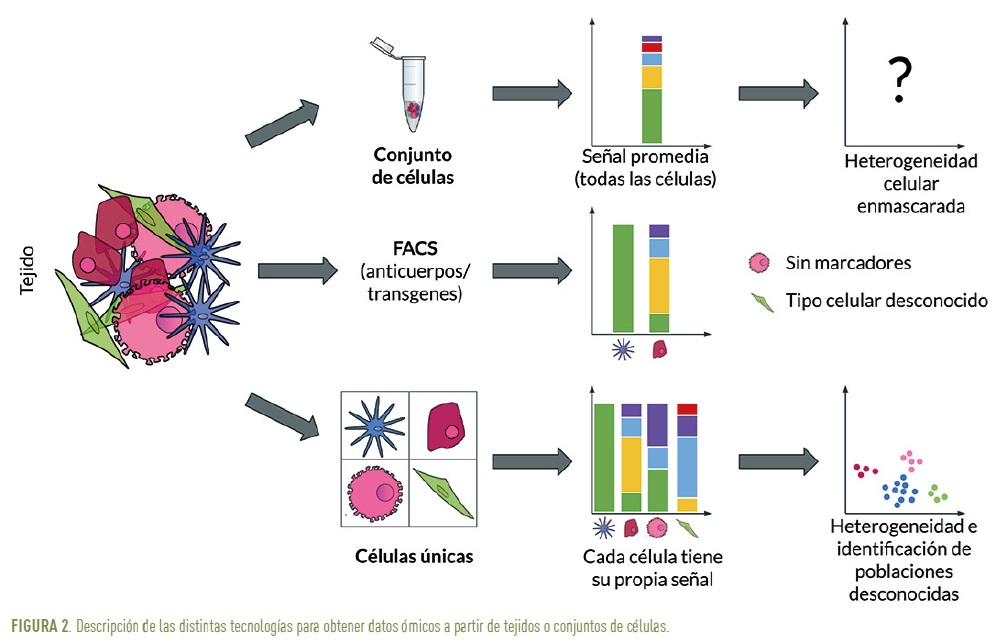
Históricamente, el estudio de las diferentes ómicas se ha realizado a partir de un conjunto de células, por ejemplo, un trozo de tejido o varios islotes pancreáticos. Así pues, la señal obtenida será la suma de la señal presente en todas las células y, en el caso de ser una población diversa, la heterogeneidad celular quedará enmascarada (Figura 1).

Existen técnicas que permiten identificar y separar un tipo celular de interés. Una de ellas es la citometría de flujo activada por fluorescencia (FACS por sus siglas en inglés), que permite contar, clasificar y analizar células individualmente en una mezcla compleja, utilizando transgenes o anticuerpos marcados con fluorocromos que se unen a proteínas específicas en la superficie celular. Uno de los inconvenientes de esta técnica es que se necesita un conocimiento previo de marcadores que permitan identificar el tipo celular de interés. Por tanto, no será posible seleccionar tipos celulares para los que no se conozcan marcadores o identificar nuevos tipos celulares no descritos anteriormente.
En este contexto, la posibilidad de usar tecnologías de célula única (single cell) permitirá caracterizar de forma independiente cada una de las células presentes en un tejido, permitiendo así analizar el grado de heterogeneidad e identificar nuevos tipos o estados celulares, sin necesidad de depender de marcadores o de conocimiento previo (Figura 2).
EN 2009 SE CONSIGUIÓ ANALIZAR EL TRANSCRIPTOMA DE UNA SOLA CÉLULA POR PRIMERA VEZ
Tipos de estrategias para obtener células únicas
En 2009 se consiguió analizar el transcriptoma de una sola célula por primera vez (1). A partir de este momento, el reto se convirtió en desarrollar estrategias para poder obtener esta información en un número cada vez mayor de células. Durante la última década, las tecnologías de célula única han ido cambiando y evolucionando, permitiendo desde la caracterización de solo unas pocas células al estudio de la composición celular de organismos completos (2). Actualmente, se suelen analizar desde decenas de miles de células hasta cientos de miles en el caso de los estudios más grandes.
En este sentido, se han desarrollado distintos tipos de estrategias para conseguir separar o identificar las células de forma individual. Cada estrategia tiene diferentes grados de sensibilidad y permite caracterizar diferentes números de células (3). Las distintas estrategias se pueden clasificar en tres grupos, en función de si están basadas en la separación de células únicas mediante: 1) tubos, microfluidos, placas o chips; 2) gotas; 3) identificación por códigos o índices moleculares.
Separación mediante tubos, microfluidos, placas o chips. En este primer grupo, cada célula se distribuye en pocillos o tubos individuales, donde se realiza la preparación del conjunto de moléculas que se pretende identificar, que conocemos como librerías. En cada una de ellas, se añade un identificador molecular para cada una de las moléculas provenientes de una misma célula. Esta tecnología permite analizar pocas células, pero consigue librerías más complejas, es decir, un mayor número de moléculas por célula.
Separación mediante gotas (droplets en inglés). En esta tecnología la suspensión de células individuales se mezcla con un aceite y una solución de reactivos e identificadores moleculares en un dispositivo microfluídico que crea gotas diminutas, cada una conteniendo una sola célula y los reactivos necesarios para realizar el análisis. Estas gotas actúan como compartimentos aislados, permitiendo realizar reacciones específicas dentro de cada gota sin interferencias de otras células. Permite identificar un gran número de células en paralelo (hasta 10,000). Esta solución, comercializada por 10X Genomics, es la más popular y utilizada actualmente.
Separación mediante identificación por códigos o índices moleculares. Esta tecnología está basada en la realización de varias rondas en las que se reparten las células en varios pocillos y se añaden identificadores moleculares. Posteriormente, las células vuelven a juntarse y se redistribuyen, añadiendo otro identificador de nuevo. Así pues, cada célula podrá ser identificada por la combinación de identificadores añadidos en cada ronda. Esta tecnología también permite obtener un gran número de células, en función del número de rondas realizadas.
LAS TÉCNICAS DE “CÉLULA ÚNICA” PERMITEN ESTUDIAR CADA CÉLULA QUE EXISTE EN UN ISLOTE PANCREÁTICO DE FORMA INDIVIDUAL
Datos ómicos que podemos obtener a partir de células únicas
Las estrategias de célula única inicialmente fueron desarrolladas para estudiar el transcriptoma de las células. Ésta sigue siendo la aplicación mayoritaria y más común de esta tecnología, pero existen otras ómicas que se pueden analizar con las estrategias descritas anteriormente (Figura 3).
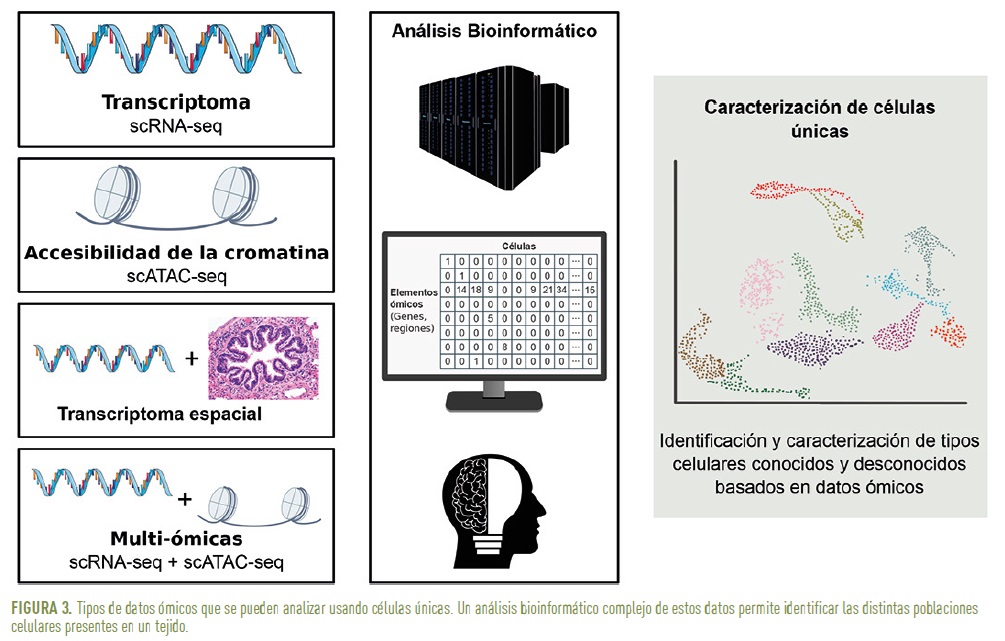
Transcriptoma (scRNA-seq). Permite identificar los transcritos (ARN) presentes en una célula. Esta tecnología facilita la clasificación y caracterización de células a nivel molecular, al revelar patrones específicos de expresión génica. Esto permite identificar cambios en la expresión génica que reflejan la funcionalidad y el estado de las células, permitiendo una comprensión más profunda de su papel en procesos biológicos y enfermedades.
Accesibilidad de la cromatina (scATAC-seq). En el núcleo celular, el ADN está empaquetado en cromatina alrededor de las histonas. Las regiones activas del genoma son accesibles a la maquinaria enzimática encargada de la transcripción, mientras que las inactivas permanecen empaquetadas. Determinar qué regiones de la cromatina están accesibles permite identificar las zonas activas del genoma, específicas para cada tipo celular. Las técnicas para identificar estas regiones se basan en su mayor sensibilidad a la rotura mediante unas enzimas conocidas como transposasas. Dado que hay muchas regiones accesibles por célula, el número de moléculas obtenidas es demasiado elevado como para permitir la secuenciación de todas ellas, por lo que el porcentaje de fragmentos obtenidos en cada célula no es muy elevado.
Célula única espacial (spatial single cell). Recientemente, también se han desarrollado técnicas que permiten añadir información sobre la localización espacial de las células, además de caracterizar su expresión génica. Esta técnica de célula única especial combina el análisis de células individuales con la preservación de la ubicación espacial original de las células dentro de un tejido. Mediante el uso tecnologías de imagen que capturan la información sobre las células en su contexto tisular, proporciona una visión detallada de cómo las células interaccionan y se distribuyen en el tejido. Esto facilita la comprensión de la heterogeneidad celular y las interacciones microambientales, un aspecto muy relevante precisamente en el estudio del ataque inmunológico durante la DT1.
Multi-ómica (multiome). Existen tecnologías que permiten obtener dos o más ómicas de cada una de las células analizadas. La más frecuente es la combinación de scRNA-seq y scATAC-seq. Esta técnica se basa en el aislamiento de núcleos y posterior caracterización de los transcritos y regiones accesibles en cada una de las células estudiadas.
Además de las técnicas mencionadas anteriormente, existen muchas otras para caracterizar diversas ómicas en células individuales, aunque no están tan estandarizadas. Entre ellas se incluyen el estudio del proteoma, metiloma, modificaciones histónicas y la estructura tridimensional de la cromatina.

Análisis de datos de célula única
El análisis de datos de células únicas presenta una complejidad significativa debido al uso de herramientas bioinformáticas avanzadas que permiten procesar y analizar grandes volúmenes de datos. La gran cantidad de información generada requiere el uso de recursos computacionales especializados, como clusters de alta capacidad. Esta abundancia de datos permite la aplicación de inteligencia artificial y técnicas de aprendizaje automático para identificar patrones y realizar predicciones. Sin embargo, el principal desafío radica en la interpretación de estos datos, ya que se genera más información de la que actualmente somos capaces de analizar y comprender.
Impacto de las tecnologías de célula única en el estudio de la DT1
Desde el desarrollo de estas tecnologías, se han aplicado al estudio de diversas patologías, incluyendo la DT1. Dado que la DT1 es una enfermedad multifactorial que implica muchos tipos celulares distintos, el estudio de células únicas permite descubrir nuevos mecanismos implicados en su desarrollo.
La secuenciación de ARN de célula única (scRNA-seq) de células mononucleares de sangre periférica (PBMCs) de pacientes con DT1 ha revelado una desregulación de múltiples tipos de células inmunitarias, incluyendo las células T CD4+ y CD8+, B, NK y células mieloides (4). Este análisis ha caracterizado una actividad diferencial de los reguladores transcripcionales en subconjuntos específicos de células inmunitarias, proporcionando información sobre los mecanismos moleculares de la disfunción inmunitaria en la DT1.
Aunque el sistema inmunitario es el principal responsable de la muerte de las células beta en la DT1, las células beta y otros tipos celulares del páncreas también juegan un papel clave en desarrollo de la enfermedad (5,6). Dado que el páncreas, y especialmente los islotes de Langerhans, contienen muchos tipos celulares distintos, las tecnologías de célula única son cruciales para caracterizarlos. Recientemente, un estudio analizó alrededor de 80,000 células de islotes pancreáticos mediante scRNA-seq y otros millones de células mediante citometría y técnicas de imagen, revelando que un subconjunto de células ductales exocrinas adquiere un perfil similar al de células dendríticas tolerogénicas en donantes con DT1 (7). Además, el análisis de células únicas ha permitido el uso de modelos de aprendizaje automático para predecir la progresión de la DT1 (8), ofreciendo oportunidades para intervenciones tempranas.
En resumen, estos hallazgos no sólo profundizan nuestra comprensión de los mecanismos patogénicos de la DT1, sino que también tienen el potencial de transformar las estrategias de predicción, prevención y tratamiento de esta enfermedad tan compleja. La identificación de estados celulares específicos y cambios epigenéticos ofrece nuevas oportunidades para desarrollar terapias dirigidas que pueden complementarse con otros tratamientos, mejorando los resultados clínicos para los pacientes con DT1.
Conclusiones
Las nuevas técnicas de célula única han revolucionado el estudio de la genómica de la DT1. Estas metodologías son poderosas, ya que permiten caracterizar la heterogeneidad celular con una resolución sin precedentes. Sin embargo, presentan limitaciones como su elevado coste y la necesidad de infraestructura bioinformática avanzada. Además, se requiere de un conocimiento especializado para la correcta interpretación de los resultados. A pesar de estos desafíos, estos avances mejoran nuestra comprensión de la patogénesis de la DT1 y abren nuevas vías para el desarrollo de terapias personalizadas y estrategias de prevención más efectivas.
Referencias
- Tang, F. et al. mRNA-Seq whole-transcriptome analysis of a single cell. Nat. Methods 6, 377–382 (2009).
- Svensson, V., Vento-Tormo, R. & Teichmann, S. A. Exponential scaling of single-cell RNA-seq in the past decade. Nat. Protoc. 13, 599–604 (2018).
- Preissl, S., Kyle J Gaulton, Ren, B. & Ren, B. Characterizing cis-regulatory elements using single-cell epigenomics. Nat. Rev. Genet. (2022) doi:10.1038/s41576-022-00509-1.
- Honardoost, M. A. et al. Systematic immune cell dysregulation and molecular subtypes revealed by single-cell RNA-seq of subjects with type 1 diabetes. Genome Med. 16, 45 (2024).
- Ramos-Rodríguez, M. et al. The impact of proinflammatory cytokines on the β-cell regulatory landscape provides insights into the genetics of type 1 diabetes. Nat. Genet. 51, 1588–1595 (2019).
- Chiou, J. et al. Interpreting type 1 diabetes risk with genetics and single-cell epigenomics. Nature 594, 398–402 (2021).
- Fasolino, M. et al. Single-cell multi-omics analysis of human pancreatic islets reveals novel cellular states in type 1 diabetes. Nat. Metab. 4, 284–299 (2022).
- Patil, A. R. et al. Modeling type 1 diabetes progression using machine learning and single-cell transcriptomic measurements in human islets. Cell Rep. Med. 5, 101535 (2024).







